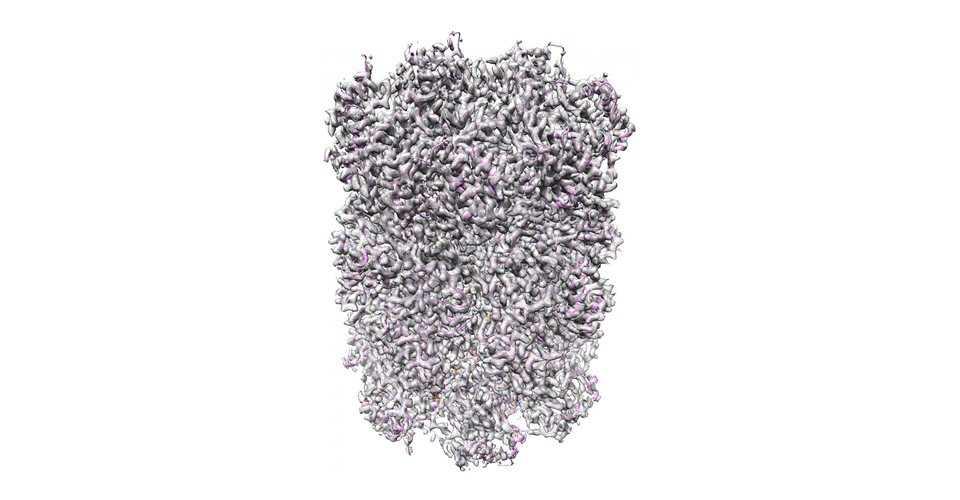Pesquisadores da Universidade de Toronto (no câmpus Scarborough) desenvolveram um conjunto de algoritmos de aprendizado de máquina (machine learning) capaz de gerar estruturas 3D em nanoescala de moléculas de proteína.
Espera-se que esses novos algoritmos revolucionem o desenvolvimento de novas terapias farmacológicas para uma diversidade de doenças, podendo inclusive explicar melhor o funcionamento da vida no nível atômico.
Um novo conjunto de algoritmos de aprendizado de máquina pode gerar estruturas 3D em nanoescala de moléculas complexas de proteína como este complexo mapa de proteassoma refinado para 2,8 Angstroms (0,28 nanômetros) em 70 min com 49,954 imagens de partículas (crédito: Structura Biotechnology Inc.)
O medicamento ideal
Os fármacos, uma vez que estão dentro do corpo, se ligam a uma molécula específica de proteína, mudando a forma 3D dessa proteína, e alterando assim a maneira como o medicamento funciona.
O medicamento ideal seria projetado em um formato que só se ligaria a uma proteína específica (ou grupo de proteínas) que estivesse envolvida em uma determinada doença, eliminando assim os efeitos colaterais que ocorrem quando os fármacos se ligam a outras proteínas no corpo.
Uma grande limitação computacional
Como as proteínas são muito pequenas – cerca de 1 a 100 nanômetros – elas não podem ser vistas diretamente sem o uso de técnicas sofisticadas como a crio-microscopia eletrônica (crio-EM). A técnica crio-EM permite a obtenção de imagens de alta resolução a partir de amostras biológicas preservadas em gelo amorfo.
Um desafio computacional é ter a correta estrutura 3D de alta resolução a partir dessas imagens em 2D.
Com as técnicas existentes leva-se vários dias ou até semanas para gerar uma estrutura 3D em um cluster de computadores, exigindo até 500.000 horas de CPU, segundo os pesquisadores. Além disso, as atuais técnicas muitas vezes geram estruturas incorretas, a não ser que um especialista forneça uma estimativa precisa da molécula que está sendo estudada.

Ali Punjani (apontando o dedo na tela do computador), doutorando na Universidade de Toronto, diz que a habilidade de determinar as estruturas 3D das proteínas é um fator crítico para entender como elas funcionam e como vão responder às terapias farmacológicas. (foto de Ken Jones)
Alta velocidade com os algoritmos
É aí que entra o novo conjunto de algoritmos*. Ele reconstrói as estruturas 3D de moléculas de proteína usando essas imagens.
Nossa abordagem resolve alguns dos principais problemas em termos de velocidade e número de estruturas que se pode determinar” – Professor David Fleet, presidente do Departamento de Ciências Computacionais e Matemáticas da Universidade de Toronto Scarborough.
Assim, os novos algoritmos podem ajudar significativamente o desenvolvimento de novos fármacos porque proporcionam um jeito mais rápido e eficiente para se chegar à estrutura correta das proteínas.
Essa nova abordagem, chamada cryoSPARC, e desenvolvida pela Structura Biotechnology Inc. (startup da equipe), elimina a necessidade de um conhecimento prévio e pode fazer em minutos os cálculos em um único computador, usando um pacote de software acelerado por GPU (unidade de processamento gráfico), de acordo com os pesquisadores.
A pesquisa foi publicada em 06/02/17 na revista Nature Methods e recebeu o financiamento do Conselho de Pesquisa de Ciências Naturais e Engenharia do Canadá (NSERC). A nova plataforma de crio-EM deles já está sendo usada em laboratórios na América do Norte.